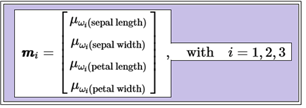
5-22 Revision of Linear Discriminant Analysis To Iris Flowers Dataset III
Fisher 교수와 Anderson 교수가 함께 완성한 Iris 즉 붓꽃 데이터 베이스에 대한 LDA formulation 및 코딩을 고려해 보자. 물론 그들이 1936년도에 데이터베이스를 완성하고 통계학 분야의 천재로 알려진 Fisher 교수가 Classification을 위한 LDA 기법을 창안한 것이 사실이지만 당시에는 지금과 같은 컴퓨터 코딩이 불가능했던 시절이었기에 과연 어디까지 했었는지 궁금증을 자아낸다.
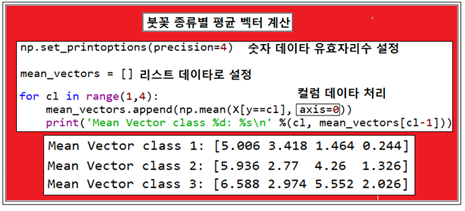
LDA 첫 단계로서 3가지 붓꽃 데이터 샘플 종류별 즉 class 라벨별로 즉 3개의 평균 벡터를 구하자.
mean_vectors 라는 변수 명을 부여한 평균 벡터는 리스트형 데이터 구조를 취하는 것으로 설정하자. 리스트형 데이터 구조를 표현할 때에 위에서처럼 컬럼형으로 매트릭스처럼 표현하지만 리스트 문법 특성 상 row 형 데이터로 봐도 무방하다. 아래의 Mean Vector class 들의 출력 결과를 보면 알 수 있을 것이다.
붓꽃 샘플 데이터는 4개씩 150개로 구성되어 있기 때문에 axis=0은 컬럼 방향으로 평균값 계산을 의미한다.
두 번째 단계에서는 3가지 붓꽃 종류 각각 별로 Scatter 매트릭스에 해당하는 Within-class scatter 매트릭스를 계산하자.

각 클래스 별 50개의 붓꽃 샘플 데이터들은 각각 꽃받침과 꽃잎의 길이와 폭 즉 4개의 독립적인 데이터로 이루어져 있다. 즉 4X50 매트릭스와 50X4 매트릭스를 곱하면 4X4 대칭형 매트릭스가 얻어진다. 개별 붓꽃 샘플 데이터를 4X1 어레이로 취급한다는 의미이다.
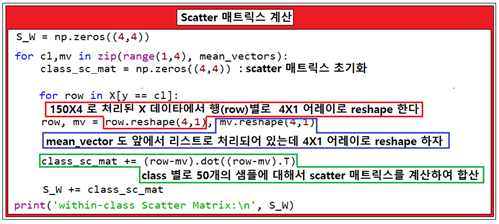
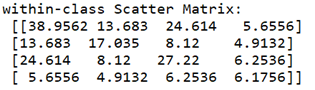
앞의 수치예제에서는 class 별 Scatter 매트릭스를 계산 후 샘플수로 나누었는데 여기서는 샘플수로 나누지 않았다는 점에 유의하자. 샘플수를 고려하여 나누어 주는 경우는 Scatter라고 하지 않고 covariance 라고 하며 unbias 된 경우 반드시 class 별로 (샘플수-1) 로 나누어 주어야 한다.
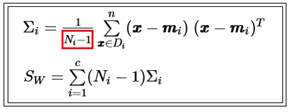
한편 붓꽃 데이터 베이스는 3가지 class의 부분집합으로 이루어져 있으므로 각 class 별 (샘플수-1) 처리를 해 주어야 이를 사용하여 계산한 합성분산 값과 전체 150개로 이루어진 샘플(population)에서 직접 계산한 전체분산 값이 같아지게 된다. 즉 covariance 값을 계산하여 처리하려면 다음과 같이 (샘플수-1) 만큼 곱하도록 한다.
Within-class scatter 매트릭스에서 covariance 가 아닌 Scatter 방식으로 계산하기 때문에 Between-class scatter 매트릭스에서도 covariance가 아니므로 (샘플수-1)이 아닌 샘플수 자체를 곱하도록 한다.

전체 평균값 및 각 class 별 샘플 수를 계산한다. 아울러 각 class 별 평균에서 전체 평균을 뺀 후 역행렬과 곱하여 누적하면 4X4 대칭형 매트릭스가 얻어진다.
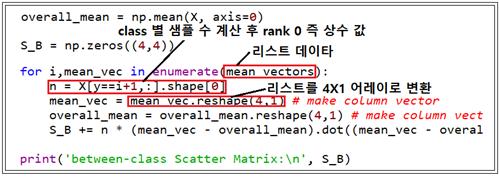

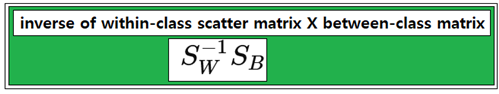
Within –class scatter 매트릭스의 역행렬과 Between-class scatter 행렬을 곱한 후 eigenvalue 문제로 정식화 하도록 한다.
Fisher의 Linear Discriminant 조건은 다음과 같다.
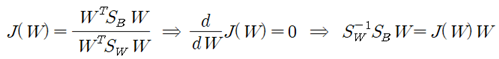
이 식에서 특정한 W 값에서 위 등식을 만족하는 조건을 적용하여 eigenvalue 방정식을 유도하자.
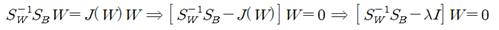
numpy 와 lapack 를 사용하여 eigenvalue 와 eigenvector를 계산하자.
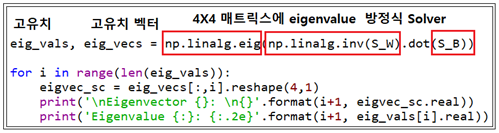
얻어진 4개의 eigenvalue 값들이 가지게 되는 정보 또는 의미를 파악해 보자.

eigenvalue 와 eigenvector는 선형변환 결과에 대한 정보를 제공하는데 특히 eigenvector는 변환의 방향을 지시하며 eigenvalue는 eigenvector의 축척으로 사용이 가능하다.
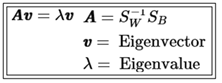
계산된 eigenvalue와 eigenvector를 대입하여 만족 여부를 체크해 본다. 대입해서 처리된 결과가 성공적이면 ‘ok’를 출력한다.

이미 계산된 eigenvalue 중에서 어느 것이 가장 유용한 정보를 제공할지 알아보자. 일단 크기순으로 처리하도록 한다. 물론 3번과 4번의 eigenvalue 값은 0.0 이므로 고려 대상에서 제외시키면 결국 4X2 변환 매트릭스가 남는 셈이다.
LDA 이론에서 주어진 데이터를 보다 낮은 차원 낮은 공간으로 투영함으로 인해 class 별 Classification 이 용이해질 수 있다는 점을 지적하였다. 여기서는 150개로 이루어진 즉 150X4 크기의 X 데이터와 4X4 로 계산된 eigenvector 중에서 eigenvalue 가 실질적으로 0.0 인 2개의 컬럼을 배제한 4X2 eigenvector를 사용하여 선형변환을 하도록 한다. 선형 변환의 결과는 운 좋게도 아래와 같이 평면상에 작도가 가능한 150X2 매트릭스로 얻어질 것이다.
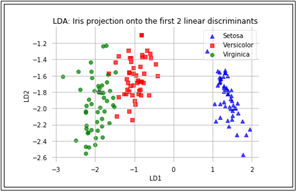
붓꽃 데이터의 class를 결정하기 위해 꽃받침과 꽃잎의 길이와 폭을 파라메터로 사용함에 아무런 무리가 없어 보인다. 하지만 Linear Discriminant Analysis 에 의하면 4차원 데이터 구조에서 3차원이 아닌 2차원 데이터 구조로 변환된다는 사실은 결국 붓꽃 데이터는 평면상에서 작도가 가능한 2개의 독립적인 파라메터로 기술된다는 점이다.
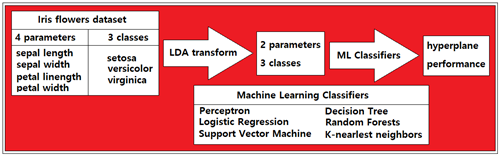
따라서 이 2개의 좌표로 표현이 가능한 변환된 붓꽃 데이터는 수많은 머신 러닝의 Classification 알고리듬 적용이 가능해진다. 적용해볼만한 Classification 알고리듬으로는 scikit-learn 라이브러리의 지원이 가능한 퍼셉트론, Logistic Regression, SVM, Decision Tree, Random Forests, KNN을 들 수 있다.
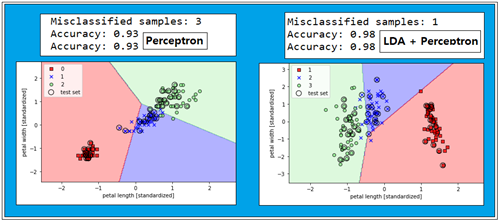
그 중 가장 기본적인 퍼셉트론에 의해 Classification 작업을 해보자. 학습 횟수는 40회, learning rate은 0.1 로 둔다. 70% 학습 30% 테스트 조건하에서 LDA 처리를 하지 않은 퍼셉트론 처리의 93%에 비해 놀랍게도 98%가 나온다. 하지만 퍼셉트론은 선형분리(linearly separable) 가능한 경우에 특화 되어 있어 더 이상의 정밀도 증가는 불가능해 보인다.

단순 퍼셉트론보다 보다 정교한 Classifiers를 사용할 경우 즉 OvR(One versus Rest) Logistic Regression기법에서 Overfitting을 감안하여 inverse regularization 파라메터 C=10.0일 경우에 한해서 100%가 나오지만 나머지 SVM, Decision Tree, Random Forests, KNN 에서는 98%가 한계이다. 이 점에 관해서는 첨부한 코드를 실행해 보면 알 수 있을 것이다.
이어서 LDA+Classifier 기법 적용 결과를 살펴보기로 한다.
#accuracy_test_01.py
from sklearn import version as sklearn_version
from sklearn import datasets
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import Perceptron
from sklearn.metrics import accuracy_score
from matplotlib.colors import ListedColormap
import matplotlib.pyplot as plt
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.linear_model import SGDClassifier
from sklearn.tree import DecisionTreeClassifier
from sklearn.tree import export_graphviz
from sklearn.ensemble import RandomForestClassifier
from sklearn.neighbors import KNeighborsClassifier
#Loading the Iris dataset from scikit-learn. Here, the third column represents the petal length, and the fourth column the petal width of the flower samples. The classes are already converted to integer labels where 0=Iris-Setosa, 1=Iris-Versicolor, 2=Iris-Virginica.
iris = datasets.load_iris()
X = iris.data[:, [2, 3]]
y = iris.target
print('Class labels:', np.unique(y))
#Splitting data into 70% training and 30% test data:
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.3, random_state=1, stratify=y)
print('Labels counts in y:', np.bincount(y))
print('Labels counts in y_train:', np.bincount(y_train))
print('Labels counts in y_test:', np.bincount(y_test))
#Standardizing the features:
sc = StandardScaler()
sc.fit(X_train)
X_train_std = sc.transform(X_train)
X_test_std = sc.transform(X_test)
#Training a perceptron via scikit-learn
ppn = Perceptron(n_iter=40, eta0=0.1, random_state=1)
ppn.fit(X_train_std, y_train)
y_pred = ppn.predict(X_test_std)
print('\nPerceptron')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % ppn.score(X_test_std, y_test))
def plot_decision_regions(X, y, classifier, test_idx=None, resolution=0.02):
# setup marker generator and color map
markers = ('s', 'x', 'o', '^', 'v')
colors = ('red', 'blue', 'lightgreen', 'gray', 'cyan')
cmap = ListedColormap(colors[:len(np.unique(y))])
# plot the decision surface
x1_min, x1_max = X[:, 0].min() - 1, X[:, 0].max() + 1
x2_min, x2_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution),
np.arange(x2_min, x2_max, resolution))
Z = classifier.predict(np.array([xx1.ravel(), xx2.ravel()]).T)
Z = Z.reshape(xx1.shape)
plt.contourf(xx1, xx2, Z, alpha=0.3, cmap=cmap)
plt.xlim(xx1.min(), xx1.max())
plt.ylim(xx2.min(), xx2.max())
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x=X[y == cl, 0],
y=X[y == cl, 1],
alpha=0.8,
c=colors[idx],
marker=markers[idx],
label=cl,
edgecolor='black')
# highlight test samples
if test_idx:
# plot all samples
X_test, y_test = X[test_idx, :], y[test_idx]
plt.scatter(X_test[:, 0],
X_test[:, 1],
c='',
edgecolor='black',
alpha=1.0,
linewidth=1,
marker='o',
s=100,
label='test set')
#Training a perceptron model using the standardized training data:
X_combined_std = np.vstack((X_train_std, X_test_std))
y_combined = np.hstack((y_train, y_test))
plot_decision_regions(X=X_combined_std, y=y_combined,
classifier=ppn, test_idx=range(105, 150))
plt.xlabel('petal length [standardized]')
plt.ylabel('petal width [standardized]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()
#LogisticRegressionGD is possible for only binary classification
#Training a logistic regression model with scikit-learn
#C=100 produces 1 sample misclassified->C=10 100%
lr = LogisticRegression(C=10.0, random_state=1)
lr.fit(X_train_std, y_train)
y_pred = lr.predict(X_test_std)
print('\nLogisticRegression:C=10.0 :OvR')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % lr.score(X_test_std, y_test))
plot_decision_regions(X_combined_std, y_combined,
classifier=lr, test_idx=range(105, 150))
plt.xlabel('petal length [standardized]')
plt.ylabel('petal width [standardized]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()
#Maximum margin classification with support vector machines
#Regardkess of C values, we have 1 misclassification
svm = SVC(kernel='linear', C=10.0, random_state=1)
svm.fit(X_train_std, y_train)
y_pred = svm.predict(X_test_std)
print('SVM kernel=linear:C=10.0')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % svm.score(X_test_std, y_test))
plot_decision_regions(X_combined_std,
y_combined,
classifier=svm,
test_idx=range(105, 150))
plt.xlabel('petal length [standardized]')
plt.ylabel('petal width [standardized]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()
#Alternative implementations in scikit-learn
#ppn = SGDClassifier(loss='perceptron', n_iter=1000)
#lr = SGDClassifier(loss='log', n_iter=1000)
#svm = SGDClassifier(loss='hinge', n_iter=1000)
#Using the kernel trick to find separating hyperplanes in higher dimensional space
svm = SVC(kernel='rbf', random_state=1, gamma=0.2, C=1.0)
svm.fit(X_train_std, y_train)
y_pred = svm.predict(X_test_std)
print('SVM kernel=rbf:gamma=0.2 C=10.0')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % svm.score(X_test_std, y_test))
plot_decision_regions(X_combined_std, y_combined,
classifier=svm, test_idx=range(105, 150))
plt.xlabel('petal length [standardized]')
plt.ylabel('petal width [standardized]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()
svm = SVC(kernel='rbf', random_state=1, gamma=10.0, C=1.0)
svm.fit(X_train_std, y_train)
y_pred = svm.predict(X_test_std)
print('SVM kernel=rbf:gamma=10.0 C=1.0')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % svm.score(X_test_std, y_test))
plot_decision_regions(X_combined_std, y_combined,
classifier=svm, test_idx=range(105, 150))
plt.xlabel('petal length [standardized]')
plt.ylabel('petal width [standardized]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()
#Decision tree learning
tree = DecisionTreeClassifier(criterion='gini',
max_depth=None,
random_state=1)
tree.fit(X_train, y_train)
y_pred = svm.predict(X_test_std)
print('\nDecision Tree')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % svm.score(X_test_std, y_test))
X_combined = np.vstack((X_train, X_test))
y_combined = np.hstack((y_train, y_test))
plot_decision_regions(X_combined, y_combined,
classifier=tree, test_idx=range(105, 150))
plt.xlabel('petal length [cm]')
plt.ylabel('petal width [cm]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()
#Combining weak to strong learners via random forests
forest = RandomForestClassifier(criterion='gini',
n_estimators=25,
random_state=1,
n_jobs=2)
forest.fit(X_train, y_train)
y_pred = svm.predict(X_test_std)
print('\nRandom Forests')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % svm.score(X_test_std, y_test))
plot_decision_regions(X_combined, y_combined,
classifier=forest, test_idx=range(105, 150))
plt.xlabel('petal length [cm]')
plt.ylabel('petal width [cm]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()
#K-nearest neighbors - a lazy learning algorithm
knn = KNeighborsClassifier(n_neighbors=5,
p=2,
metric='minkowski')
knn.fit(X_train_std, y_train)
y_pred = svm.predict(X_test_std)
print('\nKNN')
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % svm.score(X_test_std, y_test))
plot_decision_regions(X_combined_std, y_combined,
classifier=knn, test_idx=range(105, 150))
plt.xlabel('petal length [standardized]')
plt.ylabel('petal width [standardized]')
plt.legend(loc='upper left')
plt.tight_layout()
