1-19 GaussianNB Classification
GaussianNB Classifier 를 사용해 보기 전에 5-0 Bayesian Rule 과 기초 확률론내용을 사전에 살펴보도록 하자. GaussianNB 는 Naive Baesian 알고리듬의 특수한 경우이다. 즉 학습과 테스트를 위한 샘플들이 연속적인 변수일 때 사용하는 알고리듬으로서 Scikit-learn의 상당 수 알고리듬들이 인식율을 높이기 위해 붓꽃 데이터의 feature 값들을 연속 변수로 취급하여 표준화 하였다.
1-4 표준화(Normalization)된 Iris 데이터 Adaline 적용에 따른 개선
https://steemit.com/kr/@codingart/1-4-normalization-adaline
현재 Scikit-learn 라이브러리 모듈인 GaussianNB()를 사용하기 때문에 그 내부코드를 알기는 어렵지만 그 원리 상 붓꽃 데이터들이 3종류의 class를 가진다면 각 종별로는 정규분포로 가정하는데 종들 간에 겹치는 부분이 충분히 있을 수 있으므로 그림에서처럼 회색의 이 겹치는 부분을 최소화해야 할 필요가 있다.
accuracy 테스트 결과는 여타의 classifier처럼 98% 수준이며 Classification 결과 표준화 영향으로 인한 곡선 형상이 고스란히 남아 있다.
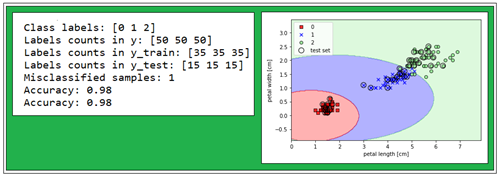
첨부된 코드를 다운받아 실행해 보자.
#GaussianNB_01.py
from sklearn import version as sklearn_version
from sklearn import datasets
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from sklearn.preprocessing import StandardScaler
from matplotlib.colors import ListedColormap
import matplotlib.pyplot as plt
from sklearn.naive_bayes import GaussianNB
#Loading the Iris dataset from scikit-learn. Here, the third column represents the petal length, and the fourth column the petal width of the flower samples. The classes are already converted to integer labels where 0=Iris-Setosa, 1=Iris-Versicolor, 2=Iris-Virginica.
iris = datasets.load_iris()
X = iris.data[:, [2, 3]]
y = iris.target
print('Class labels:', np.unique(y))
#Splitting data into 70% training and 30% test data:
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.3, random_state=1, stratify=y)
print('Labels counts in y:', np.bincount(y))
print('Labels counts in y_train:', np.bincount(y_train))
print('Labels counts in y_test:', np.bincount(y_test))
#Standardizing the features:
sc = StandardScaler()
sc.fit(X_train)
X_train_std = sc.transform(X_train)
X_test_std = sc.transform(X_test)
def plot_decision_regions(X, y, classifier, test_idx=None, resolution=0.02):
# setup marker generator and color map
markers = ('s', 'x', 'o', '^', 'v')
colors = ('red', 'blue', 'lightgreen', 'gray', 'cyan')
cmap = ListedColormap(colors[:len(np.unique(y))])
# plot the decision surface
x1_min, x1_max = X[:, 0].min() - 1, X[:, 0].max() + 1
x2_min, x2_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution),
np.arange(x2_min, x2_max, resolution))
Z = classifier.predict(np.array([xx1.ravel(), xx2.ravel()]).T)
Z = Z.reshape(xx1.shape)
plt.contourf(xx1, xx2, Z, alpha=0.3, cmap=cmap)
plt.xlim(xx1.min(), xx1.max())
plt.ylim(xx2.min(), xx2.max())
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x=X[y == cl, 0],
y=X[y == cl, 1],
alpha=0.8,
c=colors[idx],
marker=markers[idx],
label=cl,
edgecolor='black')
# highlight test samples
if test_idx:
# plot all samples
X_test, y_test = X[test_idx, :], y[test_idx]
plt.scatter(X_test[:, 0],
X_test[:, 1],
c='',
edgecolor='black',
alpha=1.0,
linewidth=1,
marker='o',
s=100,
label='test set')
#Decision tree learning
#tree = DecisionTreeClassifier(criterion='gini', max_depth=4, random_state=1)
#tree.fit(X_train, y_train)
gnb = GaussianNB()
gnb.fit(X_train, y_train)
#y_pred = tree.predict(X_test)
y_pred = gnb.predict(X_test)
print('Misclassified samples: %d' % (y_test != y_pred).sum())
print('Accuracy: %.2f' % accuracy_score(y_test, y_pred))
print('Accuracy: %.2f' % gnb.score(X_test, y_test))
X_combined = np.vstack((X_train, X_test))
y_combined = np.hstack((y_train, y_test))
plot_decision_regions(X_combined, y_combined,
classifier=gnb, test_idx=range(105, 150))
plt.xlabel('petal length [cm]')
plt.ylabel('petal width [cm]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()