바이러스학 실험 – Viral dsRNA isolation
CF-11 column purification은 virus가 감염된 filamentous fungus, Botrytis cinerea 등에서 빠르게 dsRNA를 분리할 수 있는 방법으로, CF11 cellulose fiber powder를 resin으로 사용하여 dsRNA를 핵산들 속에서 선택적으로 분리할 수 있는 방법입니다. Whatmantm 사의 powder는 alcohol 존재 시 RNA, DNA가 binding하게 합니다.
실험 재료로는 CF-11 column(Whatmantm), 1 tube, 2 tube, TSE buffer, ethanol를 준비하였습니다.

먼저 Tube setting을 시작하였습니다. 냉동고에 저장되어 있던 2 tube (2X ethanol)을 centrifugation (12K, 10min)를 꺼내어 상등액을 버리고, 뒤집어서 dry 시킵니다. TSE 2ml를 넣어서 녹인 후 100㎕를 따서 3 tube로 labeling 후 냉동보관합니다. 냉동고에 있던 1 tube와 전 과정 후 남은 2 tube용액을 purification 합니다.
먼저 수화(with TSE buffer)를 overnight합니다. 각 조당 2개씩 column – transfer pipet의 끝을 잘라서 구멍을 크게 만듭니다. packaging in column 작업 후, Equilibrium : TSE로 5ml 합니다. 그 후 TSE+20% ethanol 로 Equilibrium을 한 번 더 해줍니다. 이 후 sample loading을 해주는데 20% ethanol을 넣어서 해줍니다. 이후 TSE와 ethanol을 이용하여 ssRNA, 단백질 조각이 다 씻겨 내려갈 수 있도록 Washing작업을 해줍니다. Elution 하여 tube에 모은 후 TSE를 제거하고, ds RNA를 모읍니다. 이 용액들을 침전시킨 후 흔들어 냉동실에 1시간 보관하면 침전이 완료됩니다. 이 후 모든 실험이 끝나면 nano drop을 통하여 측정합니다.
2 tube에는 ethanol이 DNA 침전을 하여서 nano-drop으로 정량하였을 때, A260에서 A280사이에서 높은 흡광도를 가지는 것을 확인할 수 있었습니다. Whatmantm 사의 powder를 사용할 때에는 washing 과정에서 20% alcohol이 RNA, DNA가 binding하게 하는 역할을 하므로 TSE buffer와 섞어서 같이 써주어야 하며, Elution 과정에서는 alcohol이 없는 TSE buffer를 사용하여 DNA를 elution 시킬 수 있습니다.
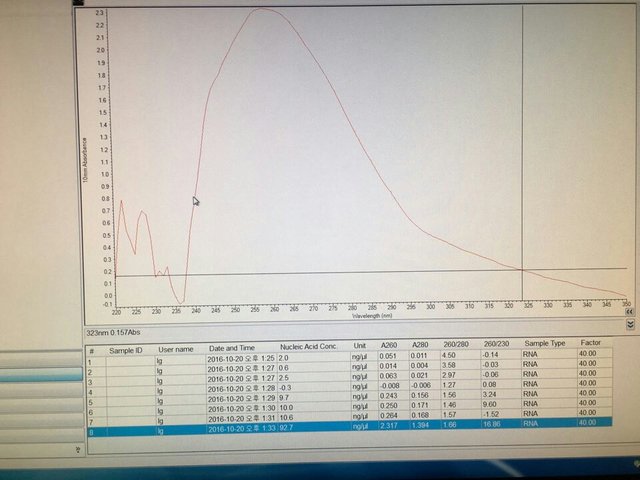
Nano drop을 할 때에는 TE buffer를 blank로 잡아서 Sample의 DNA 흡광도에서 TE buffer의 흡광도를 빼주어 온전히 DNA만의 흡광도를 구해내야 합니다.
260/280 ratio of 1.66... not all that pure maybe still around 60% protein contamination (dependent on the sequence of your RNA). I see you didn't do a phenol chloroform extraction, might improve the purity by including that step, prior to your ethanol precipitation :D